L’ibridazione genomica comparativa su microarray (
Array - Comparative Genomic Hybridization o Array-CGH) è una tecnica sviluppata per identificare identificare anomalie cromosomiche di tipo numerico (aneuploidie) a carico dei 22 autosomi (cromosomi dal nr. 1 al nr. 22) e dei cromosomi sessuali (X e Y), o anche variazioni (Variazioni del numero di copie – CNV) del contenuto di piccole porzioni cromosomiche, come duplicazioni/amplificazioni (presenza di copie in eccesso di segmenti di DNA), o delezioni (perdite di porzioni di genoma). Queste anomalie del DNA possono essere la causa di diverse patologie quali, ad esempio, sindromi malformative, ritardo mentale, autismo, epilessia e tumori.
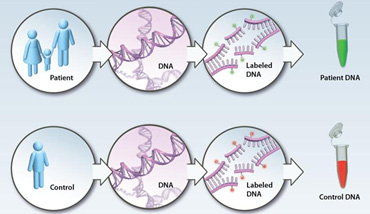
Il principio su cui si basa la tecnica dell’Array CGH è la comparazione quantitativa del DNA in esame o
DNA test (estratto dalle cellule fetali, in caso di diagnosi prenatale, o dal prelievo ematico del paziente, in caso di diagnosi post-natale) e del DNA genomico di riferimento proveniente da un soggetto sano (
reference DNA).
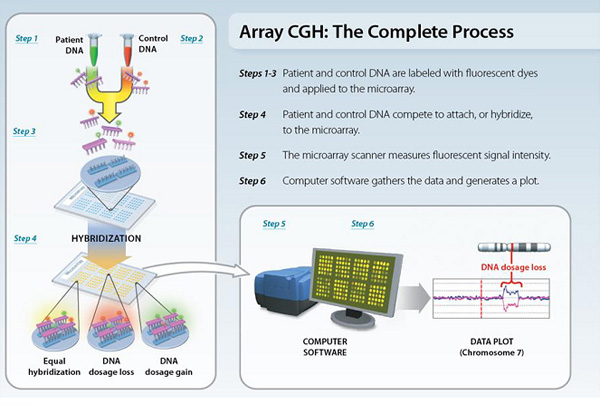
Durante il processo analitico questi DNA sono marcati in maniera differenziale con molecole fluorescenti (generalmente si utilizza un fluorocromo rosso il DNA test ed un fluorocromo verde per il reference DNA) e, successivamente, vengono mescolati in parti uguali e fatti incubare (Ibridazione) su un microarray, costituito da un supporto di vetro la cui superficie è coperta di frammenti di DNA, noti come sonde o cloni. Ognuno di questi cloni rappresenta una specifica regione del genoma umano, fino a ricomprendere l’intero assetto cromosomico umano. Tanto più è elevato il numero di cloni maggiore è l'efficacia dell'array nell'identificazione delle variazioni del numero di copie. corrispondenti a piccole porzione di ciascun cromosoma. Il potere risolutivo della piattaforma utilizzata può variare in funzione della densità e della tipologia delle sonde utilizzate; attualmente per scopi diagnostici vengono impiegati array tra 1 Mb e 100 kb.
Al termine della suddetta incubazione, sia il DNA in esame che quello di controllo si legheranno ai cloni presenti sull’array. Il risultato sarà l’emissione di due distinti segnali fluorescenti le cui intensità saranno misurante a seguito di lettura degli arrays mediante un apposito strumento (scanner). Sull’immagine ottenuta verrà poi effettuata l’analisi comparativa tra le intensità di fluorescenza emesse dai due DNA e la relativa elaborazione dei dati mediante un apposito software, al fine evidenziare eventuali variazioni del numero di copie del DNA test.
In caso di assetto cromosomico normale, il rapporto tra le due emissioni è bilanciato (1:1). Qualora vi siano nel DNA in esame (fetale) delle delezioni (assenza di un cromosoma o parte di esso), il rapporto tra quest’ultimo ed il DNA di controllo sarà di 1:2 (monosomia completa o parziale). Nel caso di duplicazioni (presenza di un cromosoma soprannumerario o parte di esso) il rapporto tra il DNA embrionale e quello di controllo sarà di 2:1 (trisomia completa o parziale).